주요 차이 UPGMA와 이웃 결합 트리 사이에는 upgma가 a 라는 것입니다. 평균 연결 방법을 기반으로 한 Glomerative 계층 적 클러스터링 방법 이웃 결합 트리는 최소 진화 기준을 기반으로 한 반복 클러스터링 방법입니다. 또한, UPGMA는 뿌리가 달린 계통 발생 트리를 생성하는 반면 이웃 결합 트리 방법은 루트되지 않은 계통 발생 트리를 생성합니다. UPGMA 방법은 동일한 진화 속도를 가정하기 때문에 분기 팁이 동일하게 발생하는 반면 이웃 결합 트리 방법은 불평등 한 진화 속도를 허용하므로 분기 길이는 변화의 양에 비례합니다.
upgma (산술 평균을 가진 비가 중 쌍 그룹 메소드)와 이웃 결합 (NJ) 트리는 두 가지 유형의 알고리즘으로, 거리 매트릭스에서 계통 발생 트리를 구축합니다. 일반적으로 UPGMA는 단순하고 빠르지 만 신뢰할 수없는 방법이며 이웃 결합 트리 방법은 비교적 빠른 방법이며 UPGMA 방법과 비교할 때 더 나은 결과를 제공합니다.
주요 영역을 다루었습니다
1. upgma
- 정의, 방법, 중요성
2. 이웃에 합류하는 트리
- 정의, 방법, 중요성
3. UPGMA와 이웃 결합 트리의 유사점은 무엇입니까
- 일반적인 기능의 개요
4. UPGMA와 이웃 결합 트리의 차이점은 무엇입니까
- 주요 차이점 비교
주요 용어
응집 클러스터링 방법, 거리 매트릭스, 이웃 결합 트리, 계통 발생 트리 
upgma
upgma (산술 평균을 가진 비가 중 쌍 그룹 메소드)는 Sokal과 Michener에 기인 한 단순하고 응집 적이며 계층 적 클러스터링 방법입니다. 뿌리와 초 미세 계통 발생 트리를 구축하는 데 가장 간단하고 빠른 방법입니다. 그러나이 방법의 주요 단점은 모든 계통에 대해 동일한 진화율에 대한 가정입니다. 이것은이 계통의 돌연변이 속도가 시간이 지남에 따라 일정하다는 것을 의미합니다. 이것을 '분자 시계 가설'이라고도합니다. 또한 나무의 모든 가지를 비슷한 거리로 생성합니다. 그러나 모든 계통에 대해 동일한 돌연변이 속도를 갖는 것이 어렵 기 때문에 실제로 UPGMA 방법은 신뢰할 수없는 나무 토폴로지를 더 자주 생성합니다.
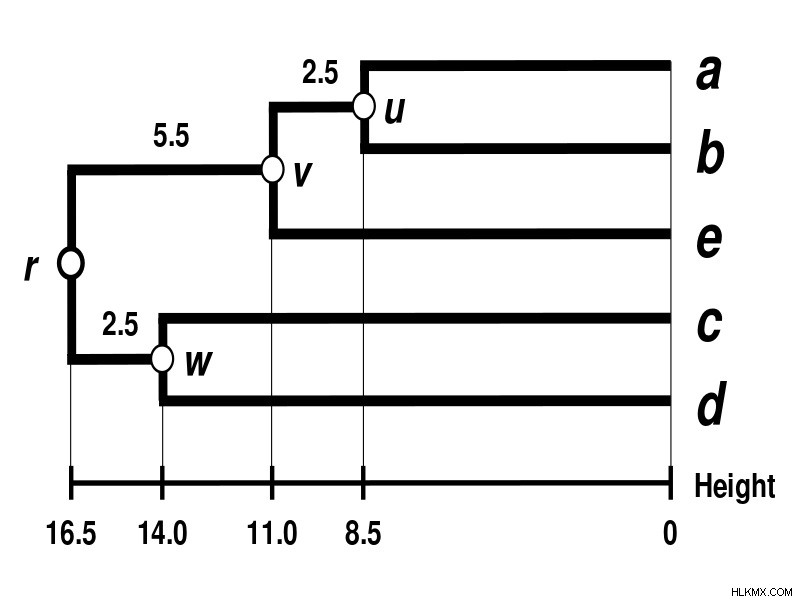
그림 1 :UPGMA 방법
또한 UPGMA 방법은 쌍별 거리의 매트릭스로 시작합니다. 처음에는 각 종이 그 자체로 클러스터라고 가정합니다. 그런 다음 거리 매트릭스에서 가장 작은 거리 값으로 가장 가까운 두 클러스터를 결합합니다. 또한, 평균을 취함으로써 조인트 쌍의 거리를 다시 계산합니다. 그런 다음 알고리즘은 모든 종이 단일 클러스터에 연결될 때까지 프로세스를 반복합니다.
이웃 결합 트리
이웃 결합 (NJ) 트리 방법은 계통 발생 트리를 건설하는 데 사용되는 최신 응집 클러스터링 방법입니다. 1987 년 Naruya Saitou와 Masatoshi Nei에 의해 개발되었습니다. 또한 초소형 거리가 필요하지 않으며 별 분해 방법을 사용합니다. 또한, 이웃 결합 트리 알고리즘은 진화율의 계보의 변화를 조정합니다. 따라서 해결되지 않은 별 같은 나무로 시작합니다.
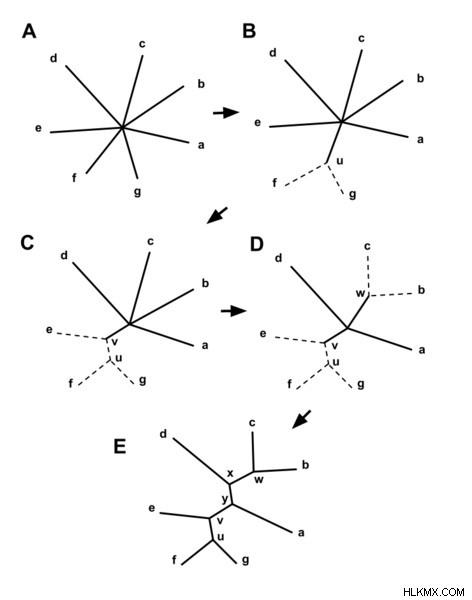
그림 2 :이웃 결합 나무 구조
이웃 결합 트리 방법에서 행렬 Q는 현재 거리에 따라 계산됩니다. 그런 다음 새로 생성 된 노드에 결합 할 거리가 가장 낮은 계보 쌍을 선택합니다. 그러나이 노드는 중앙 노드와 관련이 있습니다. 그 후 알고리즘은 각 계보에서 새 노드까지의 거리를 계산합니다. 그런 다음 각 린지에서 외부에서 새 노드까지의 거리를 계산합니다. 마지막으로, 계산 된 거리를 기반으로 결합 된 이웃을 새로운 노드로 대체합니다.
UPGMA와 이웃 결합 트리 간의 유사성
- upgma 및 neighbor-joining tree는 거리 매트릭스를 입력으로 사용하는 두 개의 알고리즘입니다. 일반적으로 거리 매트릭스는 2D 매트릭스입니다. 포인트 세트의 쌍별 거리를 포함하는 배열입니다.
- 관련 단백질 또는 DNA 서열 세트의 결과 정렬 점수는 거리 화합물의 구성을위한 측정으로 사용될 수 있습니다.
- 둘 다 응집 적 (상향식) 클러스터링 메소드입니다.
- 계산적으로 저렴한 더 빠른 방법입니다.
- 따라서 대규모 데이터 세트에 적용 할 수 있습니다.
- 또한 두 방법 모두 다른 유형의 입력과 비교할 때 더 나은 결과를 얻습니다.
- 는 단일 트리를 생성하도록 설계되었지만 때로는 하나 이상의 토폴로지를 생성하여 데이터 센터 순서를 기반으로 '혼돈 적'동작을 초래합니다.
- 부트 스트랩 값은 노드/클레이드 형성 가능성을 확인하기위한 간단한 통계 테스트입니다.
UPGMA와 이웃 결합 트리의 차이
정의
upgma는 거리 매트릭스에서 뿌리를 뿌린 계통 발생 트리를 구성하기위한 간단한 접근법을 말하면서 이웃 결합 트리는 스타 트리를 통해 부근하지 않는 계통 발생 트리를 구성하기위한 새로운 접근법을 나타냅니다.
에 의해 개발되었습니다
upgma 방법은 1958 년 Sokal과 Michener에 의해 개발되었으며 1987 년 Naruya Saitou와 Masatoshi Nei에 의해 이웃 결합 트리가 개발되었습니다.
의 중요성
UPGMA는 평균 연결 방법을 기반으로 한 응집 적 계층 적 클러스터링 방법이며 이웃 결합 트리는 최소 진화 기준을 기반으로 한 반복 클러스터링 방법입니다.
계통 발생 수의 유형
upgma 메소드는 뿌리가 많은 계통 발생 트리를 구축하는 동안 이웃 결합 트리 방법은 루트되지 않은 계통 발생 트리를 구축합니다.
거리의 유형
또한 UPGMA 알고리즘은 거리가 초소형이어야하는 반면 이웃 결합 트리 알고리즘은 거리가 중독성이어야합니다.
계통 발생 트리의 가지의 특성
upgma 메소드가 동일한 진화 속도를 가정하면 분기 팁이 동일하게 나타납니다 (루트에서 팁까지의 분기 길이). 이웃 결합 트리 방법이 불평등 한 진화 속도를 허용하므로 분기 길이는 변화의 양에 비례합니다.
속도
upgma는 이웃 결합 트리는 비교적 빠른 방법입니다.
신뢰성
또한 UPGMA는 신뢰할 수없는 방법이며 이웃 결합 트리는 더 나은 결과를 생성합니다.
결론
upgma는 진화 거리 데이터를 기반으로 계통 발생 트리를 구축하기위한 두 알고리즘 중 하나입니다. 또한, 비슷한 가지 길이를 가진 뿌리가있는 계통 발생 트리를 만듭니다. 또한 거리 행렬에서 계통 발생 트리를 구축하기위한 단순하고 빠르며 가장 신뢰할 수있는 알고리즘입니다. 반면, 이웃 결합 트리는 거리 매트릭스에서 계통 발생 트리를 만드는 데 사용되는 두 번째 방법입니다. 그러나, 그것은 분기 길이가 진화 동안 변화의 양을 반영하는 루트되지 않은 계통 발생 트리를 생성합니다. 또한이 알고리즘은 알고리즘이 비교적 덜 빠르지 만 가장 신뢰할 수있는 계통 발생 트리를 구축합니다. 따라서 UPGMA와 이웃 결합 트리의 주요 차이점은 계통 발생 트리의 특징과 알고리즘의 특징입니다.