흔들림 가설은 제 3 차 코돈 염기와 제 1 항 코돈 염기 사이의 결합이 표준 왓슨 크릭베이스 쌍을 따르지 않아, 다수의 코돈이 단일 아미노산을 코딩 할 수 있다고 설명한다.
단백질을 구성 할 수있는 20 개의 다른 아미노산이 있습니다 (희귀 아미노산을 세면 22 :셀레 노시 슈타인과 피 롤리 신). 각각의 아미노산은 리보솜, mRNA 및 TRNA의 도움으로 제조되어 단백질 생성 기계를 구성합니다. 아미노산은 목걸이의 구슬처럼 리보솜에서 다른 한 번에 첨가됩니다. 결과 '목걸이'는 폴리펩티드 사슬로 단백질로 접습니다.
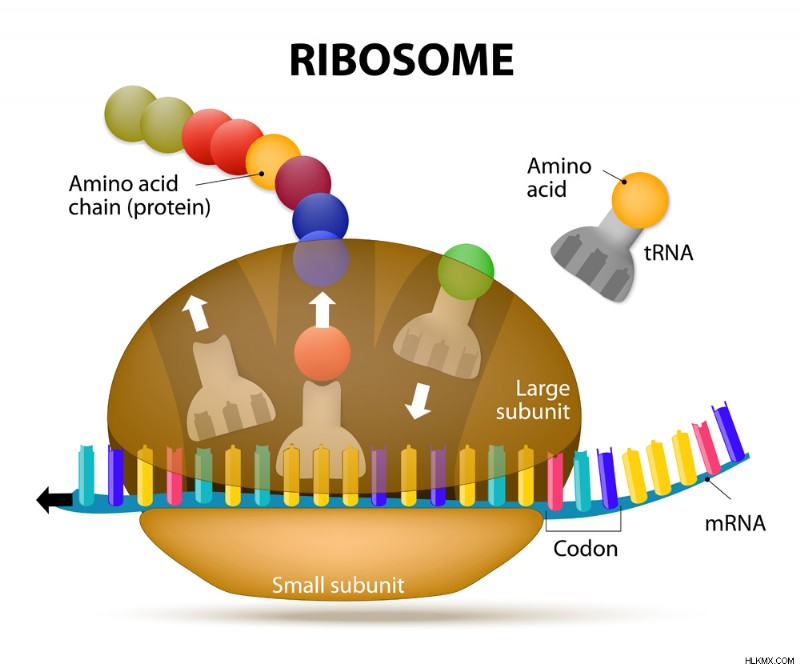
리보솜에서 단백질을 형성하는 번역 기계 (사진 신용 :DesignUA/Shutterstock)
여러 코돈이 단일 아미노산을 코딩 할 수있는 방법을 밝혀 내기 전에 관련된 플레이어에 대해 조금 더 배우겠습니다.
 Gene Expression. 세포질의 리보솜은 새로운 단백질 (Soleil Nordic) S "width ="1000 "높이 ="657 "을 합성합니다. srcset ="https://www.scienceabc.com/wp-content/uploads/2020/09/DNA-Translation-Are-part-of-gene-expression.-Ribosomes-in-the-cytoplasm-synthesize-proteil-nordics-1.jpg 10 000w, 10,000w, https://www.scienceabc.com/wp-content/uploads/2020/09/DNA-transcript-and-translation-are-part-of-the-gene-expression.-ribosomes-in-the-cytoplasm-synthesize-new-proteinssoleil-nordics --new-300x197.jpg 300w "size" 100VW, 1000px "> 중심 교리 :mRNA는 DNA에서 전사되어 핵을 리보솜으로 맡기고, 그곳에서 번역을 돕고 단백질을 초래합니다 (사진 신용 :Soleil Nordic/ShutterStock)
Gene Expression. 세포질의 리보솜은 새로운 단백질 (Soleil Nordic) S "width ="1000 "높이 ="657 "을 합성합니다. srcset ="https://www.scienceabc.com/wp-content/uploads/2020/09/DNA-Translation-Are-part-of-gene-expression.-Ribosomes-in-the-cytoplasm-synthesize-proteil-nordics-1.jpg 10 000w, 10,000w, https://www.scienceabc.com/wp-content/uploads/2020/09/DNA-transcript-and-translation-are-part-of-the-gene-expression.-ribosomes-in-the-cytoplasm-synthesize-new-proteinssoleil-nordics --new-300x197.jpg 300w "size" 100VW, 1000px "> 중심 교리 :mRNA는 DNA에서 전사되어 핵을 리보솜으로 맡기고, 그곳에서 번역을 돕고 단백질을 초래합니다 (사진 신용 :Soleil Nordic/ShutterStock)
mRNA :번역의 출발점
메신저 RNA 또는 mRNA는 단일 가닥의 RNA입니다. DNA의 정보가 복사되거나 전사되는 전사라는 과정에서 합성됩니다. mRNA 가닥으로. 이중 가닥 DNA 나선이 열리고 5 '내지 3'코딩 가닥과 동일한 mRNA 가닥이 생성됩니다. 리보 핵산 (RNA)은 티민 (t) 대신 뉴클레오티드 우리 딘 (U)을 함유하며, 이는 DNA의 mRNA와 코딩 (5 '내지 3')의 유일한 차이점이다.
.
전사 및 번역 과정 (사진 크레디트 :Becky Boone/Wikimedia Commons)
그런 다음 mRNA는 단백질 제작 기계 인 리보솜으로 나아갑니다. 리보솜은 단백질을 만들기 위해 mRNA의 정보를 읽습니다. 이것을 번역하는 것과 같은 변환이라고합니다.
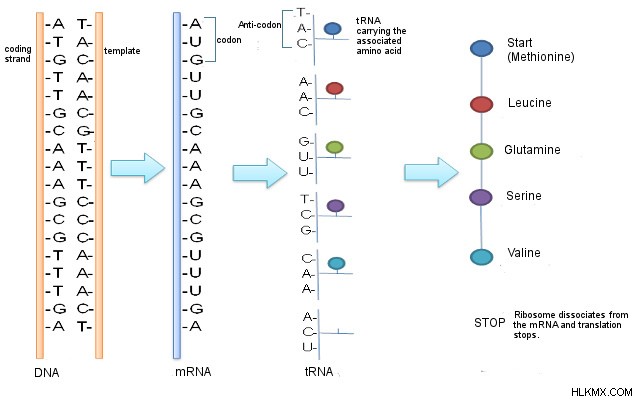
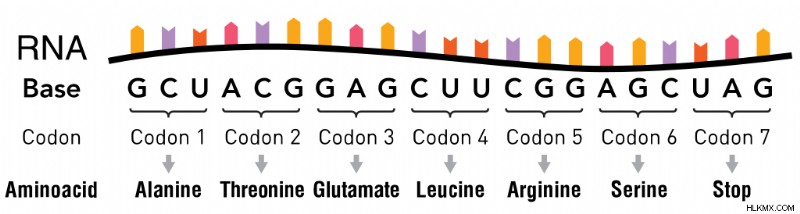
mRNA의 정보는 코돈이라는 섹션에서 한 번에 3 개의 뉴클레오티드를 읽습니다. 각 코돈은 특정 아미노산을 지정합니다.
mRNA에 존재하는 각 코돈은 코돈을 지정합니다 (사진 신용 :Thomas Splettstoesser/Wikimedia Commons)
예를 들어, 코돈 Aug는 아미노산 메티오닌 (MET)에 대한 코딩한다. Aug는 또한 단백질 정보가 시작되는 경우, START 코돈이라고 불리는 코드는 번역 과정을 시작하는 데 필요한 코드입니다. 따라서, 메티오닌은 항상 아미노산 사슬에서 첫 번째 아미노산이다. 아미노산 트립토판 (TRP)은 코돈 UGG로 표시됩니다.
번역 과정을 시작하는 시작 코돈 (사진 크레디트 :Gritsalak Karalak/Shutterstock)
코돈은 아미노산을 코딩 할뿐 아니라; 또한 번역 프로세스를 종료 할시기를 코딩합니다. 그것들은 종합적으로 정지 코돈으로 알려져 있으며 UAG, UAA 및 UGA입니다.
따라서 코돈은 아미노산을 얻는 데 필요한 3 글자 비밀번호와 비슷하며 단백질 만들기를 시작하고 멈추라는 지시가 발견되는 곳.
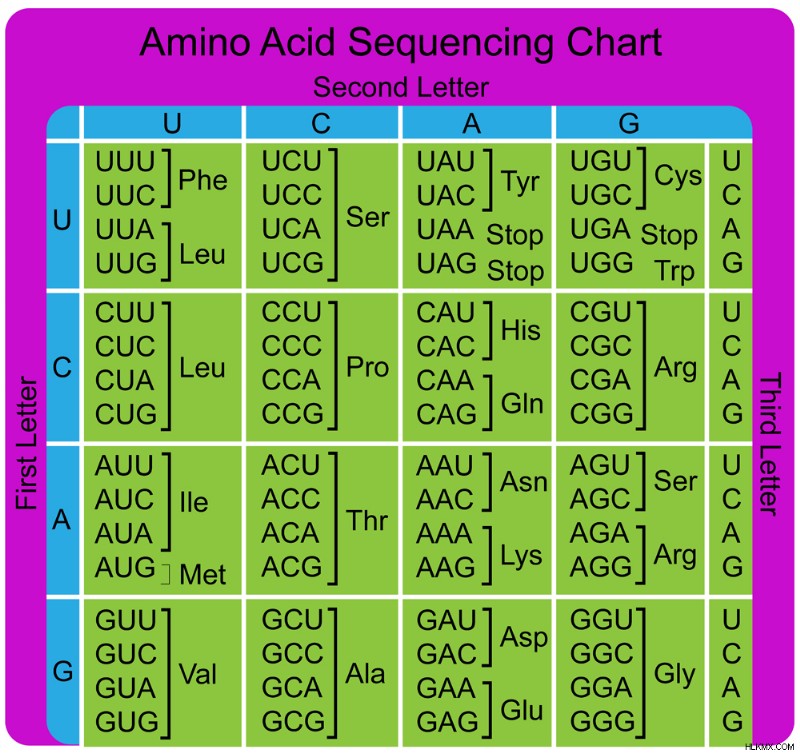
각 아미노산에 대한 코돈 (사진 크레디트 :Gstraub/Shutterstock)
trna :번역의 조립 지점
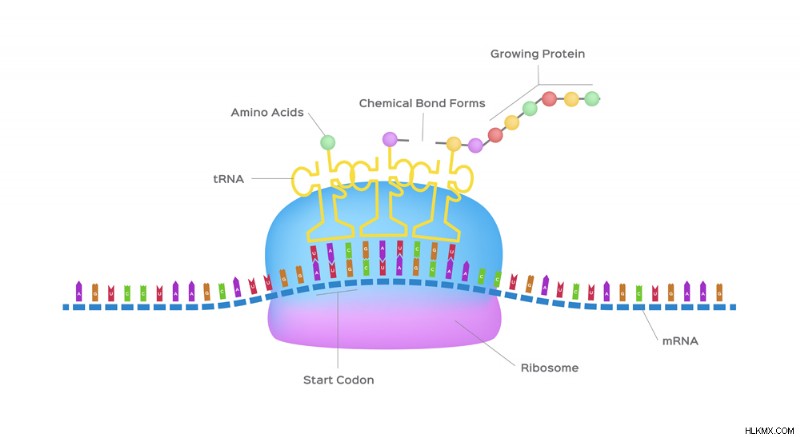
전이 RNA 또는 TRNA는 또한 단일 가닥이지만 직선 mRNA와 달리 접 힙니다. 그것은 mRNA를 해석하여 mRNA와 아미노산 사이의 물리적 연결 역할을합니다. 단백질을 만드는 동안 오른쪽 아미노산. 번역 기계가 공장 인 경우 TRNA는 지시 매뉴얼 (mRNA)을 지정된 순서로 신중하게 넣기 위해 지시 매뉴얼 (mRNA)을 해석하는 공장 노동자입니다.

TRNA는 아미노산을 조립하여 폴리펩티드 사슬 (사진 신용 :Lenam14/Wikimedia Commons)을 형성하는 공장 노동자와 같습니다.
TRNA는 한쪽 끝에 안티 코돈 루프가 있고 반대쪽 끝에 수용자 암/줄기가있는 방식으로 구성됩니다.
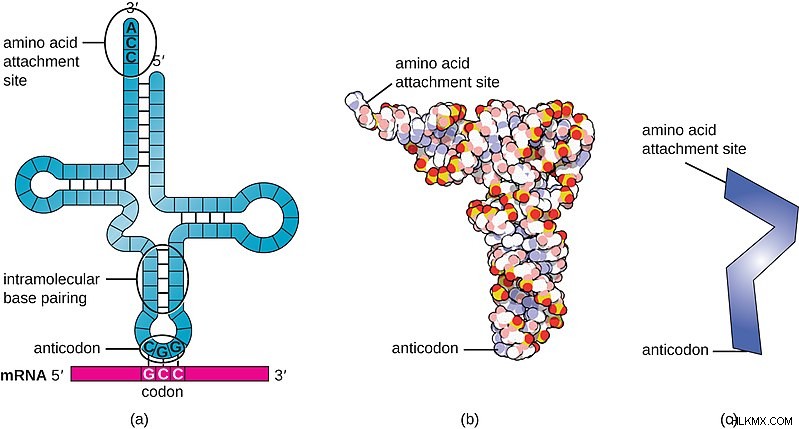
TRNA의 1 차, 2 차 및 3 차 구조 (사진 크레디트 :CNX OpenStax/Wikimedia Commons)
이름에서 알 수 있듯이, 안티 코돈 루프는 상보 적이며 mRNA 코돈에 대한 항구 평행 (3 '내지 5”)입니다. 즉, TRNA는 mRNA에 존재하는 것들에 결합하는 3 개의 뉴클레오티드로 구성된다. 따라서, 구아닌 (G) 및 시토신 (C)은 서로 결합하고 아데닌 (A) 및 우리 딘 (U)는 서로 결합한다.
따라서, 메티오닌의 코돈을 고려할 때, 그것은 항-코돈 UAG를 가진 메티오닌 tRNA를 가질 것이다.
.이 유형의 페어링은 Watson-Crick베이스 페어링으로 알려져 있습니다. 페어링은 매우 특정한 자석 사이의 매력과 같습니다. TRNA상의 항 코돈은 그것이 mRNA의 올바른 코돈에 대해 일치하도록한다. 또한 코돈이 5 '내지 3'방향으로 읽으면서 TRNA에 존재하는 안티 코돈은 3 '내지 5'방향으로 위치합니다.
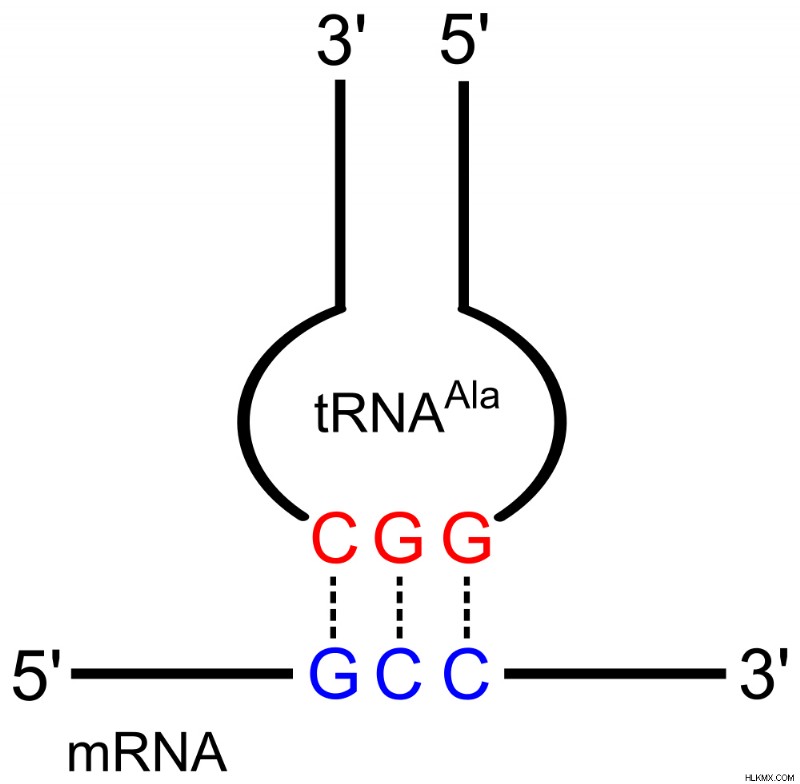
아미노산 알라닌에 대한 코돈, 항 코돈 및 TRNA (사진 신용 :Yikrazuul/Wikimedia Commons)
이는 제 1 코돈 염기가 제 3 항 안티 코돈베이스 등에 결합 함을 의미합니다. 각 아미노산은 자체 TRNA를 가지고 있으며, 코돈과 안티 코돈 사이의 염기 쌍으로 인해 폴리펩티드 사슬에 올바르게 배치합니다.
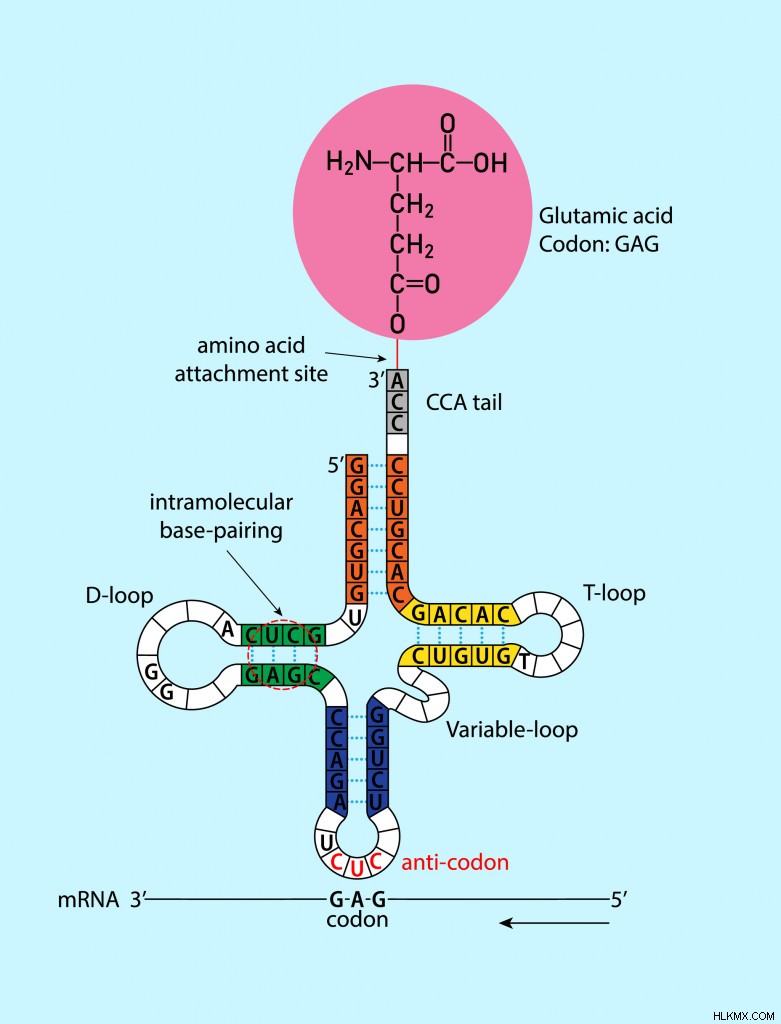
수용체 암에 글루탐산을 갖는 TRNA의 항 코돈에 결합 된 글루탐산에 대한 코돈. (사진 크레디트 :M. Patthawee/Shutterstock)
그러나 생물학의 많은 것들과 마찬가지로 이것은 조금 더 복잡합니다. 각각의 아미노산은 하나 이상의 코돈에 의해 지정 될 수있다. 또한, 코돈과 안티 코돈 사이의 기본 페어링 규칙이 모든 염기에 대해 동일하게 결합되지는 않습니다. 다음 에이 독특한 현상에 대해 더 많이 배울 것입니다!
Wobble 가설 :왜 20 개의 아미노산에 64 개의 코돈이 있는가?
우리가 알다시피, DNA는 4 개의 뉴클레오티드 염기 (A, G, T, C)로 구성됩니다. 이 글자 (기지)는 한 번에 3 개 씩 읽습니다. 즉,이 트리플렛 또는 코돈의 64 개 (4 x 4 x 4) 가능한 조합이 있음을 의미합니다.
64 개의 코돈 중 3 개는 정지 코돈이며, 이전에 언급했습니다. 이 3 개의 정지 코돈은 아미노산에 대한 코딩이 아니며 번역 과정 만 종료합니다. 따라서, 우리는 단지 20 개의 아미노산에 대해 61 개의 코돈을 남겼습니다.
유일한 논리적 옵션은 단일 아미노산이 다수의 코돈에 의해 코딩 될 수 있다는 것이다. 그러나 이론적으로는 61 개의 다른 코돈에 61 개의 다른 TRNA가 필요하지만, 그렇지 않습니다. 자연에서 50 개 미만의 코돈이 확인되었습니다.
이 관찰은 코돈과 안티 코돈 사이에 일치하는 여유가있을 수 있음을 의미합니다. Guu, Guc, Gua, Gug의 4 개의 코돈으로 코딩 된 아미노산 발린을 고려하십시오. 첫 번째베이스 만 변경하지만 첫 번째는 동일합니다.
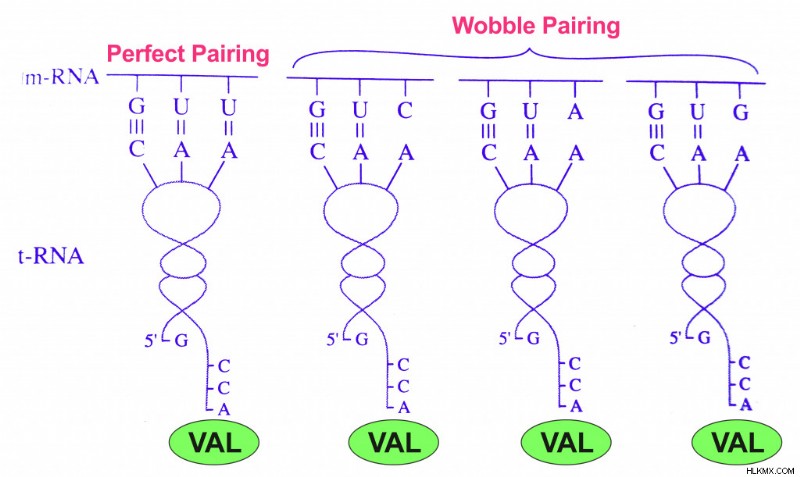
Valine의 Watson-Crick베이스 페어링 및 Wobble Base 페어링
이 차이는 코돈의 3 번째 기저에서만 관찰 된 것이 1966 년 Francis Crick (DNA 구조의 발견 자)이 제안한 'Wobble Hypothesis'로 이어졌습니다.
Wobble 가설은 Watson-Crick베이스 페어링의 규칙이 코돈의 마지막 위치에서 따르지 않았다는 것을 밝혀서 이러한 분산을 설명했습니다. 즉, 3 번째 문자 (뉴클레오티드 염기)와 항 코돈의 첫 번째 문자 사이의 유대는 비정상적인 결합 (비 A-T/ 비 G-C)을 보일 수 있습니다. 따라서‘흔들림’이 허용되었습니다.
이것은 코돈의 첫 번째와 두 번째 염기가 각각 안티 코돈의 3 번째 및 2 차 기저에 결합함으로써 Watson-Crick베이스 쌍의 규칙을 엄격히 준수했지만, 비 Watson-Crick베이스 쌍은 코돈의 3 번째 염기와 안티 코돈의 1 번째베이스 사이에 허용되었다. 따라서 일부 TRNA는 많은 다른 코돈을 인식 할 수 있습니다. 또한 유전자 코드 (단일 아미노산의 많은 코돈)에서 중복 패턴을 설명합니다.
흔들리는 가설의 규칙은 다음과 같습니다.
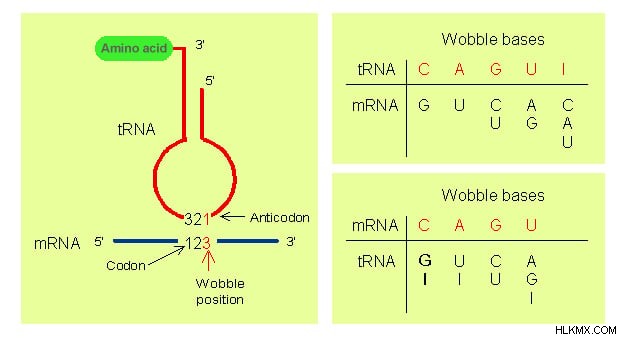
흔들리는 가설의 규칙
- 코돈의 첫 두 염기와 안티 코돈의 마지막 두 염기는 정상적인 Watson-Crick베이스 쌍을 겪습니다. 즉, 수소 결합은 아데닌 (A)와 우리 딘 (U), 구아닌 (G) 및 시토신 (C) 사이에 형성됩니다.
- 나머지 위치에 적용되는 기본 페어링 규칙 덜 적용되며 비 왓슨 크릭베이스 페어링이 발생할 수 있습니다. 그러한 페어링을 겪는베이스는 또한 흔들림베이스 쌍이라고도합니다. 이것은 TRNA의 단일 형태 (아미노산 특이 적)의 항 코돈이 mRNA에서 하나 이상의 코돈과 쌍을 이룰 수있게한다. .
- uridine (u)이 나머지 (안티 코돈의 1 기초) 위치에 존재하는 경우 아데닌 (A) 또는 구아닌 (G) 만 인식 할 수 있습니다.
- 나머지 (안티 코돈의 제 1 기저) 위치에 구아닌 (g)이 존재하면 우리 딘 (U) 또는 시토신 (c) 만 인식 할 수 있습니다.
- 변형 된 염기 이노신 (I)이 나머지 (안티 코돈의 1 기초) 위치에 존재하는 경우, 우리 딘 (U), 시토신 (C) 또는 아데닌 (a)을 인식 할 수 있습니다.
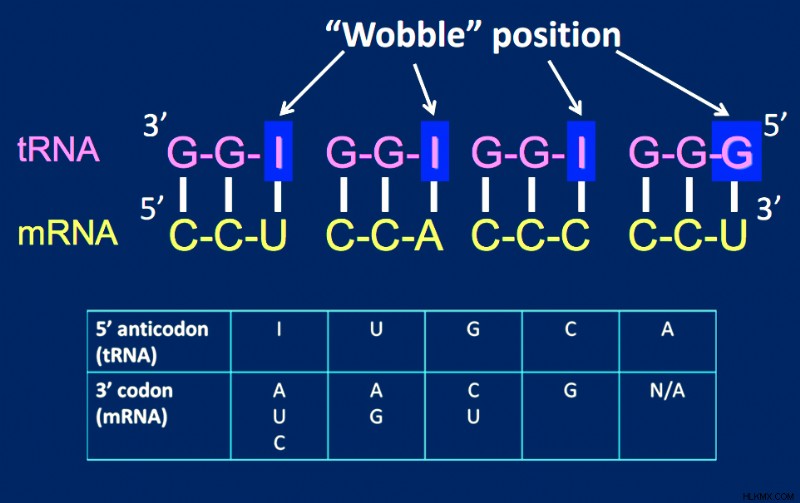
이노신은 A, U 또는 C
에 결합 할 수 있습니다
흔들림베이스 페어링의 이유는 리보솜 부분의 미끄러짐 때문입니다. 리보솜은 1 차 및 2 차 코돈 염기가 각각 3 차 및 2 차 항 코돈 염기에 상보 적인지 여부를 확인하는 메커니즘을 갖는다. 그러나, 리보솜은 TRNA 일치의 mRNA 및 1st Anticodon베이스의 3 번째 코돈 기초가 있는지 확인하는 메커니즘이 없다.
흔들림 가설의 중요성
미끄러짐이든 아니든, 흔들림 기지로 인해 번역 중에 실수가 적습니다. 예를 들어, 류신 (LEU) 코돈 CUU가 전사 동안 CUA, CUG 또는 CUC로 잘못 읽힌 경우, 코돈은 단백질 합성 동안 여전히 류신 (LEU)으로 번역됩니다.
.또한, 단백질 합성은 또한 단일 염기의 돌연변이에 대해 보호된다. 따라서, 발린 코돈 GUU의 3 번째 기저가 돌연변이되고 Guc, Gua 또는 Gug로 변경 되더라도 여전히 올바르게 번역됩니다.
유전자 코드의 Wobble 및 퇴행성은 또한 세포에 필요한 TRNA의 수를 감소시킨다. 예를 들어, 4 개의 다른 글리신 코돈의 경우 e.coli 에 존재하는 3 개의 trnas 만 있습니다. .
따라서 다수의 코돈이 단일 아미노산을 코딩 할 수 있다는 것은 사실이지만, 3 차 코돈 염기와 제 1 항 코돈베이스 사이에서 관찰되는 'Wobble'은 이러한 유연성을 가능하게한다.







